对于人类蛋白质相互作用网络的结构解析
大家好,本周为大家分享一篇发表在Nat. Struct.上的文章,Towards a structurally resolved human protein interaction network,该文章的通讯作者是瑞典斯德哥尔摩大学的Petras Kundrotas、Arne Elofsson和欧洲分子生物学实验室的Pedro Beltrao。
蛋白质-蛋白质相互作用(PPIs)的表征对于理解形成功能单位的蛋白质组和细胞生物学研究的基础是至关重要的。同时,蛋白质复合物的结构表征是理解蛋白质的功能机制、研究突变的影响和研究细胞调控过程的关键步骤。
最近,基于神经网络的方法已经被证明了准确预测单个蛋白质和蛋白质复合物的结构的能力;然而,其在大规模预测人类复杂结构中的应用尚未得到有效测试。在此,本文测试了应用AlphaFold2在预测人类蛋白质相互作用结构上的潜力和局限性,并通过实验提示了界面残基中潜在的调节机制。除此之外,本文还提供了使用预测的二元复合物来构建高阶组装的案例,以此拓展了对于人类细胞生物学的理解。
人类蛋白质相互作用的结构预测
本文基于AlphaFold2的FoldDock管道对65484对来源于HuRI与hu.MAP V.2.0数据库中实验测定的PPIs的结构进行预测。文章合并了一个pDockQ分数,该分数可以根据置信度对模型进行排序。结果显示,已知相互作用蛋白的pDockQ往往高于随机集;对于hu.MAP数据集显示出平均比HuRI数据集更高的可信度,这表明,高可信度模型集中在具有高亲和力和直接相互作用的蛋白质相互作用区域。实验表明,AlphaFold2可以预测大型复合物中直接相互作用的蛋白对的结构(图1)。
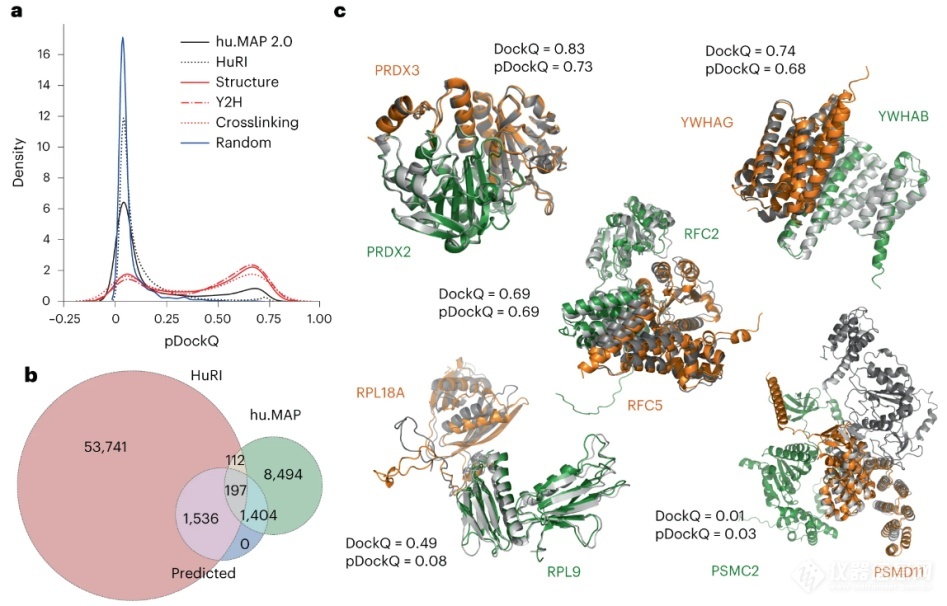
图1 | AlphaFold2复合物预测在大规模人类PPIs数据集上的应用
影响预测置信度的特征
如图1a所示,相较于HuRI和hu. MAP数据库中的蛋白质对,出现在蛋白质数据库(PDB)中的蛋白质对更加富集于高分模型部分。为了更好地理解这种差异,本文首先研究了一个由大型(>10链)异质蛋白复合物构建的额外数据集。通过实验,结果显示直接相互作用对与间接相互作用对之间pDockQ分数的差异是显著的,这表明与间接相互作用对相比,即使直接相互作用对是大型复合体的一部分,也往往能够被预测。除此之外,由于HuRI数据库中的许多蛋白质间相互作用很可能是短暂的,而AlphaFold2无法可靠地预测这种相互作用(图2)。
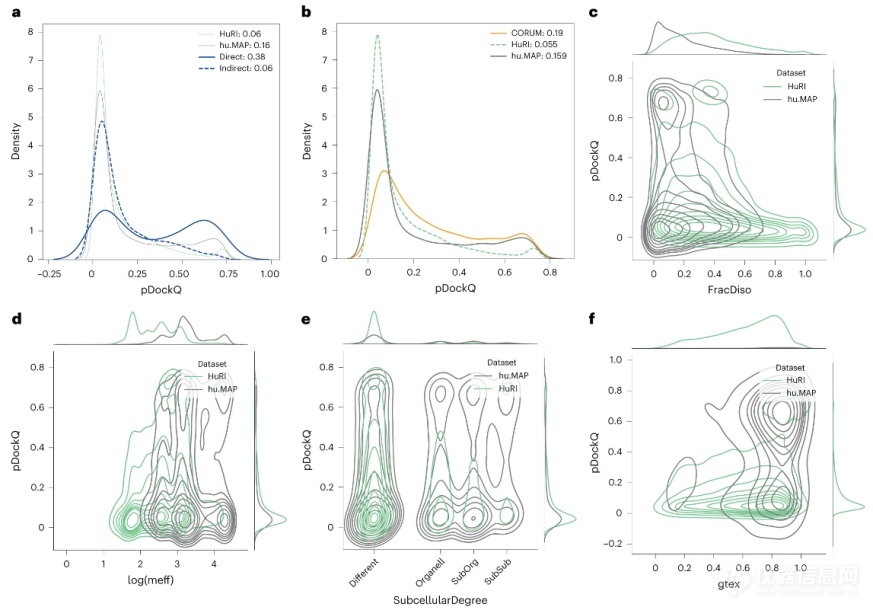
图2 | 影响预测置信度的蛋白质和相互作用特征:不同数据集的分析
预测的复合物结构在化学交联上的验证
化学交联结合质谱分析是一种识别蛋白质对中邻近的活性残基的方法,可以用来帮助确定可能的蛋白质界面。为了确定预测的复合物结构是否满足这种正交空间约束,本文获取了528对具有预测模型的蛋白质对的残基对的交联集合。在此章节中,文章提供了多个案例证明了化学交联验证的有效性(图3)。
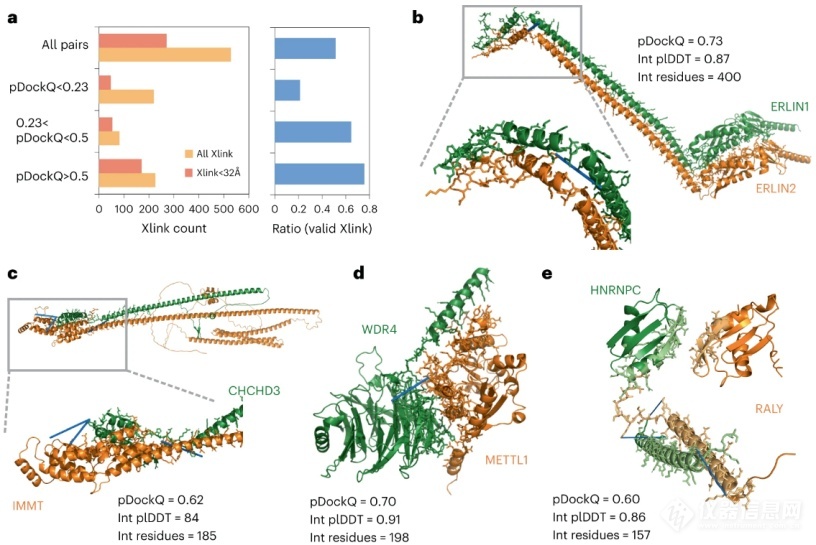
图3 | 对于预测复合物模型的化学交联支持
复合物界面上与疾病相关的错义突变
与人类疾病相关的错义突变可以通过多种机制改变蛋白质的功能,包括破坏蛋白质的稳定性、变构调节酶活性和改变PPIs。为了确定预测结构的有效性,本文汇编了一组位于界面残基上的突变,这些突变之前曾被实验测试过对于相应相互作用的影响。文章使用FoldX预测突变时结合亲和力的变化,并观察到破坏相互作用的突变强烈影响了结合的稳定性;另外,本文就在一系列生物学功能中具有界面疾病突变的蛋白质网络簇进行了举例说明(图4)。
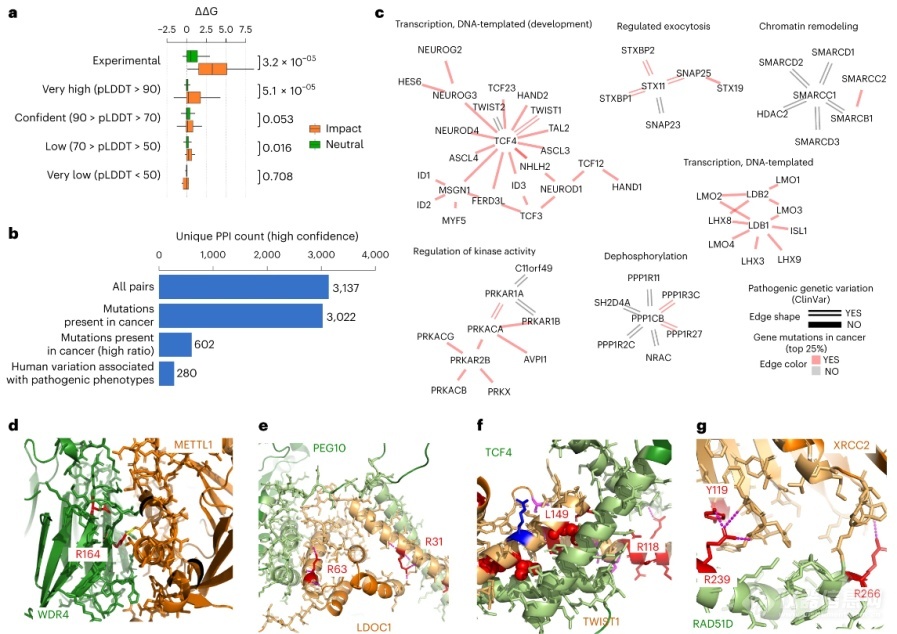
图4 | 蛋白质复合物界面残基的疾病突变
蛋白质复合物界面的磷酸化调节
蛋白质磷酸化可以通过改变修饰残基的大小和电荷来调节结合亲和力来调节蛋白质的相互作用,将磷酸化位点定位到蛋白质界面可以为它们在控制蛋白质相互作用中的功能作用产生机制假说。本文使用了最近对人类磷酸化蛋白质组26的鉴定,在高置信度模型中鉴定出了界面残基上的4,145个独特的磷酸化位点。实验表明,某些界面可能受到特定激酶和条件的协调调控。虽然不是所有界面上的磷酸位点都可能调节结合亲和力,但这一分析为特定扰动后的相互作用的潜在协调调控提供了假设(图5)。
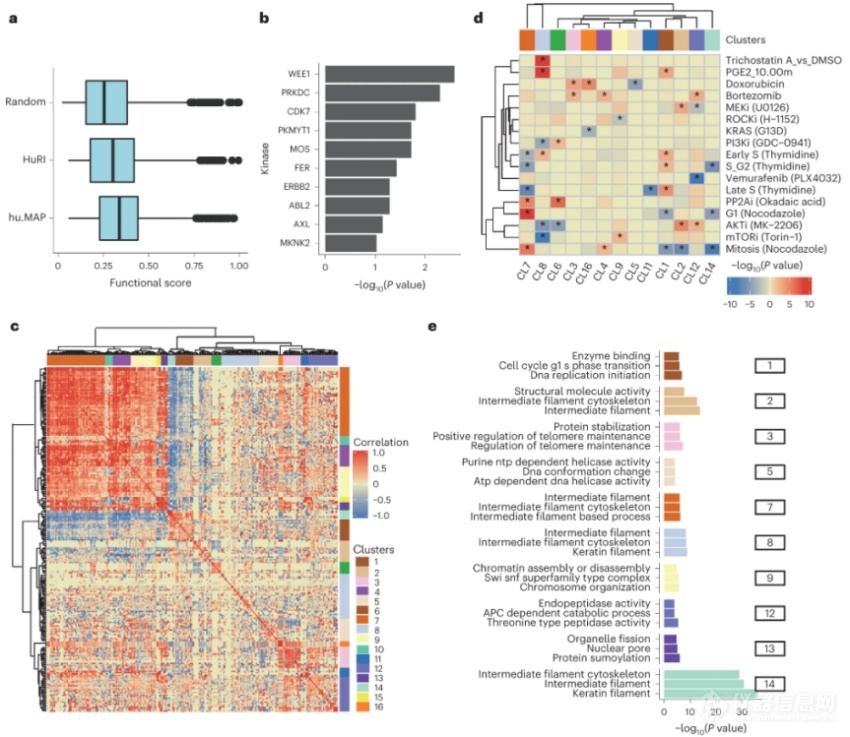
图5 | 界面残基上磷酸化位点的协同调控
来自二元蛋白质相互作用的高阶组装
蛋白质既能够同时与多个伙伴相互作用组成更大的蛋白复合物,又能够在时间和空间上分离。这也反映在文章的结构特征网络中,即蛋白质可以在群体中被发现,如蛋白质相互作用全局网络视图所示(图6)。
由于使用AlphaFold2预测更大的复合物组装可能受到计算需求的限制,文章测试了蛋白质对的结构是否可以迭代结构上对齐。文章在上述网络中覆盖的一组小的复合物上测试了这一过程,并将一个实验确定的结构与预测的模型进行对齐,展示了该过程的潜力和局限性。
受测试例子的鼓励,本文定义了一个自动化过程,通过迭代对齐生成更大的模型。总之,文章发现可以迭代地对齐相互作用的蛋白质对的结构来构建更大的组装,但同时也发现了目前限制这一过程的问题。
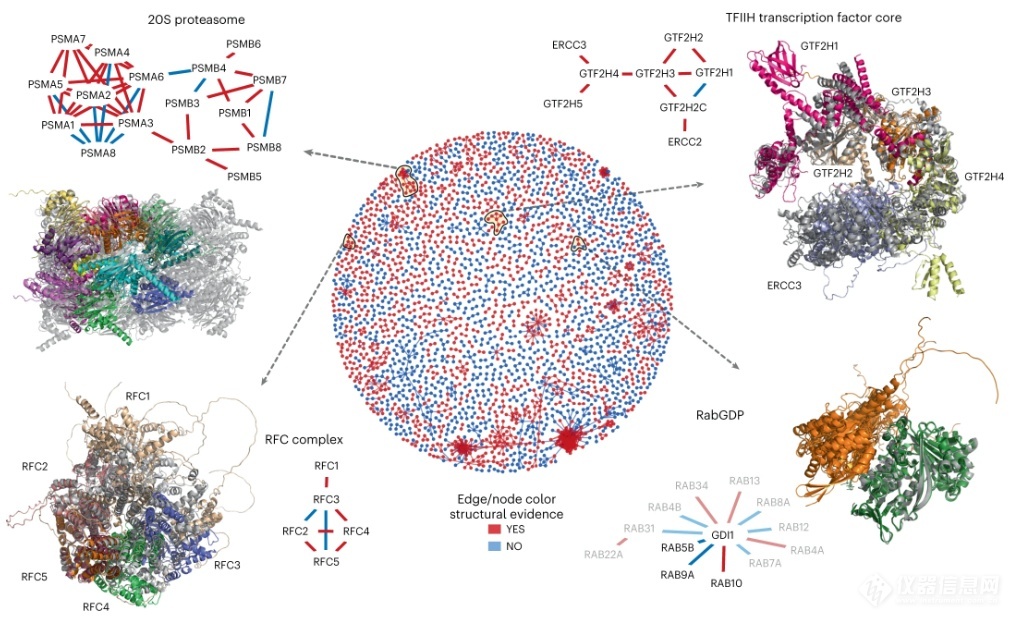
图6 | 对高阶组装的蛋白质复合物的预测
结论
本文通过一系列的实验评估了应用AlphaFold2预测已知人类PPIs的复杂结构的潜力与局限性。分析结果表明,由亲和纯化、共分馏和互补的方法组合支撑的蛋白质相互作用能够产生更高置信度的模型。文章证明,可以使用模型指标(如pDockQ评分)对高置信度模型进行排序,为大规模PPIs和稳定复合物的详细研究提供支持;而来自交联质谱实验的数据为进一步验证这些预测提供了理想的资源。
除此之外,本文用疾病突变和磷酸化数据证明了蛋白质界面的结构模型对于理解分子机制以及突变和翻译后修饰的影响至关重要;最后,文章提出了从预测的二元配合物出发构建更大的组件结构模型的想法。后续仍需要更多的工作来确定确切的化学计量学,设计方法和评分系统来构建如此更大的复杂组件,以及预测具有弱和瞬态相互作用的蛋白质之间的相互作用。
参考文献
(1) Burke DF, Bryant P, Barrio-Hernandez I, et al. Towards a structurally resolved human protein interaction network [published online ahead of print, 2023 Jan 23]. Nat Struct Mol Biol. 2023;10.1038/s41594-022-00910-8. doi:10.1038/s41594-022-00910-8
