基于碰撞活化解离技术的非变性自上而下质谱用于蛋白复合物高级结构解析
大家好,本周为大家分享一篇最近发表在 Journal of the American Chemical Society上文章,Native Top-Down Mass Spectrometry with Collisionally Activated Dissociation Yields Higher-Order Structure Information for Protein Complexes1。该文章的通讯作者是美国加利福尼亚大学洛杉矶分校的Joseph A. Loo教授。
非变性质谱(native MS,nMS)通常用于揭示蛋白及其复合物的分子量大小和化学结合计量比,但若要进一步阐明深层次的结构信息,则需要与串联质谱结合,即非变性自上而下质谱(nTDMS),通过对母离子进行二级甚至多级碎裂可获取额外的序列、翻译后修饰(PTMs)以及配体结合位点信息。此外,nTDMS能以构象敏感的方式断裂共价键,这样就可以从碎片模式推断出有关蛋白高级结构的信息。值得注意的是,使用的激活/解离方式会极大地影响得到的蛋白质高阶结构信息。
电子捕获/转移解离(ECD、ETD或ExD)和紫外光解离(UVPD)等快加热的活化方式因其能够在保留蛋白整体结构的情况下先对共价键进行断裂而被广泛应用于nTDMS分析中。而慢加热的活化方式如碰撞活化解离(CAD)会在断键前进行能量重排,导致一些较弱的非共价相互作用先发生破坏,例如:亚基的释放和展开,因此对高阶结构表征没有帮助。而此次Joseph A. Loo课题组的研究结果显示使用基于orbitrap的高能C-trap解离(HCD)同样也可以从天然蛋白复合物的中直接获得序列信息,并且碎片模式可以提供有关其气相和溶液相高阶结构信息。此外,CAD还可以生成大量的内部碎片(即不包含N-/ C-端的片段)用于揭示蛋白质复合物的高阶结构。
为了研究蛋白复合物HCD的碎裂化情况,作者比较了酵母来源的乙醇脱氢酶四聚体(ADH)在Complex-down MS (psedo-MS3)和nTDMS两种分析策略下的碎片模式。如图1所示,在Complex-down MS分析中,ADH经源内解离(ISD)释放出单个亚基,该亚基经HCD碎裂生成肽段b/y离子。而在nTDMS分析中,肽段离子则可以从复合物中直接获得。如图2(上)所示,在Complex-down MS分析中总共获得了24个b离子和18个y离子,能够实现11.8%的序列覆盖率。近乎相等数目的b、y离子表明Complex-down MS分析中释放的ADH亚基N-端和C-端均具有较高的表面可及性,即亚基发生去折叠。此外,碎片模式也揭示了N-端乙酰化、V58T突变体以及Zn2+结合位点等信息。相比之下,nTDMS分析则更反映ADH的高阶结构,如图2(下)所示,在nTDMS分析中主要检测到b离子,几乎没有亚基信号,说明b离子是直接由复合物中共价键断裂产生的。ADH的nTDMS分析共产生了60个N-端b离子和3个C-端y离子(17.6%序列覆盖率)。由HCD产生的大量N端碎片类似于ADH基于电子和光子解离技术产生的nTDMS产物。将这些片段映射到ADH的晶体结构上可以看出,N端区域比C端区域更容易暴露于溶剂,而C端区域主要形成复合物的亚基-亚基界面。ADH的碎片离子中来源亚基界面断裂的仅占8%,大部分碎裂都发生在溶剂可及的N-端。

图1 Complex-down MS和nTDMS分析流程
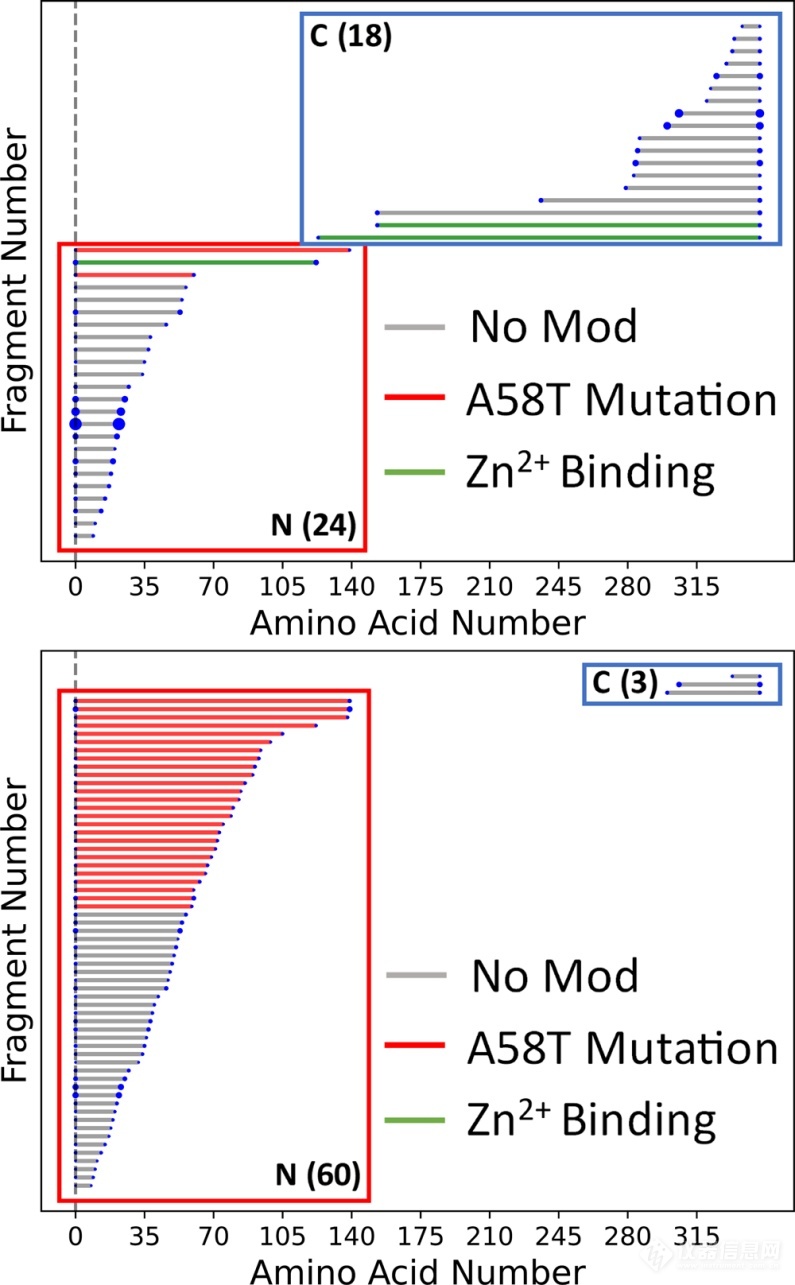
图1 Complex-down MS(上)和nTDMS(下)碎片模式比较
ADH的nTDMS分析充分展现了CAD在蛋白复合物高阶结构表征上的潜力,为了进一步验证,作者还选择了其他的蛋白复合物进行实验,如醛缩酶同源四聚体、谷胱甘肽巯基转移酶A1二聚体、肌酸激酶二聚体等。这些蛋白复合物在n-CAD-TDMS分析中都产生了与结构对应的碎片离子,说明基于CAD的nTDMS分析是具有普适性。当然也会存在一些例外,膜蛋白水通道蛋白(AqpZ)同源四聚体在nTDMS分析过程中产生了高丰度的单体亚基、二聚体、三聚体信号,这应该归因于AqpZ四聚体亚基之间的弱疏水结合界面,导致亚基的释放发生在共价键断裂之前,因此产生的b/y离子无法反映蛋白复合物的空间结构。相较而言,以盐桥为主要稳定作用的蛋白复合物,如ADH、醛缩酶等则更容易在nTDMS分析中产生肽段碎片离子。此外,基于CAD的nTDMS分析中还发现了大量的内部碎片,ADH产生的大部分内部碎片来源于溶剂可及区。尽管内部碎片难以辨认,但可以大幅度提高序列覆盖率,提供更精细的结构信息。
一个从小分子裂解衍生到大分子解离的假设是,在实验的时间尺度内,由碰撞引起的激活是完全随机化的,并以沿着最低能量途径引导碰撞诱导的解离。然而,这些假设没有考虑到熵的要求,缓慢重排可能是释放亚基所必须的,例如重新定位盐桥将一个亚基与其他亚基相连。在碰撞次数或每次碰撞能量不足以碰撞出能释放亚基的罕见构型的情况下,以释放出更小的多肽碎片(具有更少的约束) 代替重排可能具有更高的竞争性。
总之,本文展示CAD在nTDMS分析中的应用,无需基于光子或电子的活化方式,CAD可直接从蛋白复合物中获得肽段离子,并且该碎裂离子能够反映蛋白复合物的空间结构。
撰稿:刘蕊洁
编辑:李惠琳
原文:Native Top-Down Mass Spectrometry with Collisionally Activated Dissociation Yields Higher-Order Structure Information for Protein Complexes

参考文献
1. Lantz C, Wei B, Zhao B, et al. Native Top-Down Mass Spectrometry with Collisionally Activated Dissociation Yields Higher-Order Structure Information for Protein Complexes. J Am Chem Soc. 2022; 144(48): 21826-21830.
