科学家们通过“DNA汤”监测海洋健康的重大进展

研究人员为这项研究结合了MBARI开发的两个新型自主平台:长距离自主水下航行器(LRAUV)和环境样品处理器(ESP)。LRAUV是一个灵活的水下机器人,可以长时间地前往海洋的偏远地区。ESP是一个机器人的“罐中实验室”,可以过滤海水并保存电子DNA以供将来研究。通过为LRAUV配备ESP技术,研究人员可以在时间和空间上扩大海洋监测的规模。相比之下,传统的海洋eDNA采样需要在一艘昂贵的研究船上花费数周时间,而且只能在局部地区进行。像这样的技术创新正在彻底改变海洋保护工作。
“我们知道eDNA是研究海洋群落的一个非常强大的工具,但我们一直受限于使用载人研究船所能完成的工作。现在,自主技术正在帮助我们更好地利用我们的时间和资源来研究海洋的新部分,”MBARI的生物海洋学家和该论文的主要作者Kobun Truelove说。
海洋生物多样性是对海洋中个体和物种丰度的一种衡量。这种相互关联的生物马赛克--从最小的浮游生物到最大的鲸鱼--支持着食物网,产生我们呼吸的空气,并调节着我们的气候。像LRAUV和ESP这样的自主工具使MBARI的研究人员能够在海洋中保持持久的存在,并以以前不可能的方式监测敏感生态系统的变化。
“生物体随着我们的海洋和大湖区的条件变化而移动,影响到依赖这些物种的人们和经济。我们需要更便宜和更灵活的方法来大规模地监测生物多样性。这项研究提供了我们所需要的eDNA和非船员技术的协同发展,直接回应了NOAA‘Omics战略计划’中规定的优先事项,”该研究的共同作者、美国国家海洋和大气管理局(NOAA)的合作者Kelly Goodwin说。

研究背景
在这项研究中,MBARI与NOAA大西洋海洋学和气象学实验室以及华盛顿大学的研究人员合作,在蒙特利湾国家海洋保护区完成了三次考察。该团队在MBARI的三艘研究船、NOAA渔业船Reuben Lasker和MBARI的LRAUVs船队之间协调样品采集。
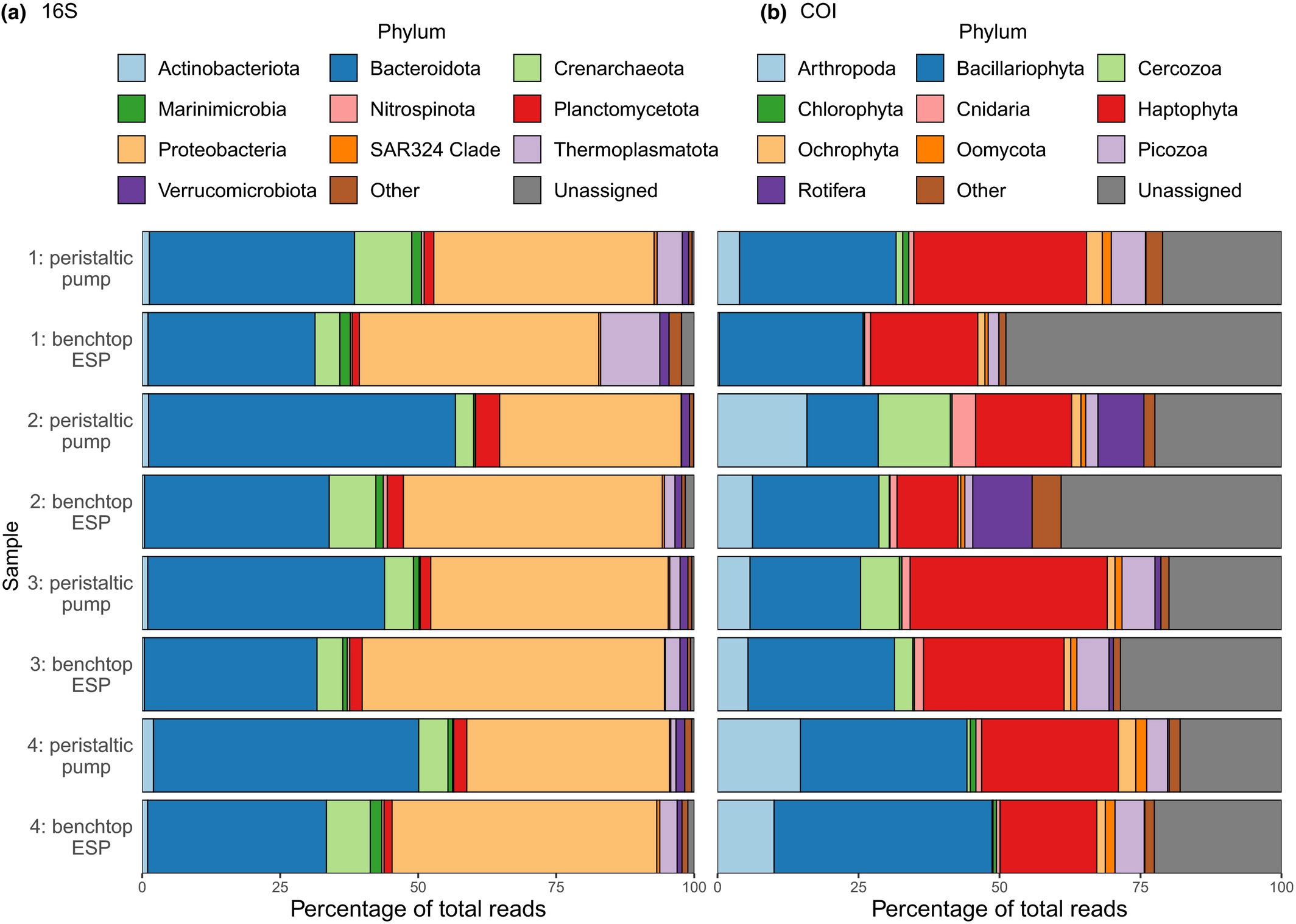
船上的团队将瓶子降到特定的深度,以收集和保存水样。同时,配备ESP的LRAUV在类似的位置和深度自主采样并保存eDNA。这些eDNA样本被送回实验室进行深入测序。
相关的生物体有共同的DNA部分,被称为基因标记。在这项研究中,研究人员用一种被称为metabarcoding的技术分析了eDNA样本。这种方法寻找短的DNA摘录,并提供样品中存在的群体的分类。这种技术对于将eDNA数据转化为生物多样性的衡量标准特别有帮助。研究人员分析了四种不同类型的基因标记,每一种都代表了食物网的一个稍微不同的层次。这些结果加在一起,产生了一个更全面的社区构成图。从研究船和自主水下航行器上收集的样本显示了类似的生物多样性模式。
Truelove指出,该研究的结果标志着在监测海洋生态系统方面迈出了令人兴奋的一步。“这项工作是关于提高eDNA研究的规模。”他说:“我们可以开始更广泛地描述海洋中的生物群落结构,而不是关注单个物种。”
“良好的数据是可持续海洋管理的基石,”MBARI高级科学家和该研究的共同作者Francisco Chavez说。“定期的环境DNA监测告诉我们谁在那里,什么在随时间变化。当涉及到了解气候变化的影响--对海洋健康的最大威胁之一--这种信息是至关重要的。”
LRAUVs能够一次行驶数周并行驶数百公里。与传统的研究船相比,它们可以在感兴趣的地区更频繁地取样,而传统的研究船通常只是不定期地访问偏远地区。自主机器人将使研究人员能够研究以前没有调查过的海洋区域。填补这些数据空白对于加强全球海洋健康至关重要。基于船舶的研究将继续在海洋学研究中发挥重要作用,但在工具包中增加新的自主技术将扩大研究、监测和资源管理的能力。最终,MBARI的研究人员设想部署一支配备ESP技术的LRAUVs舰队。
